Microbiologie naturelle volume 8 , pages1392–1396 ( 2023 ) Citer cet article
Abstrait
Au cours des deux dernières décennies, l’intérêt pour la recherche sur le microbiome humain a augmenté de façon exponentielle. Malheureusement, cette activité accrue s’est accompagnée d’un certain battage médiatique et de désinformation, qui peuvent saper les progrès et la confiance du public dans la recherche. Ici, nous mettons en lumière certains mythes et idées fausses sur le microbiome humain qui manquent de preuves solides. En présentant ces exemples, nous espérons attirer davantage l’attention sur les implications d’un dogme inexact qui s’incruste dans la littérature et sur l’importance de reconnaître les nuances lors de la description du microbiome humain complexe.
Principal
La recherche sur le microbiome humain a connu une croissance rapide au cours des deux dernières décennies et des milliers d’articles de recherche sur ce sujet sont désormais publiés chaque année. D’énormes sommes d’argent ont été dépensées pour étudier le microbiome humain en tant que cause ou solution thérapeutique potentielle à un large éventail de maladies, y compris les maladies inflammatoires de l’intestin et les conditions cardiométaboliques. Bien que vraiment passionnante, l’attention croissante portée à la recherche sur le microbiome a malheureusement également entraîné un battage médiatique et enraciné certaines idées fausses. En conséquence, de nombreuses déclarations non étayées ou insuffisamment étayées sont devenues des «faits» en raison d’une répétition constante. Certains sont plus répandus que d’autres et certains sont relativement insignifiants, mais, cumulativement, ils soulignent que la désinformation est omniprésente dans la littérature sur le microbiome humain. Compte tenu de l’importance potentielle des microbiomes humains pour la santé, il est crucial que les allégations soient fondées sur des preuves. Dans cette perspective, nous mettons en lumière les mythes et les idées fausses persistants ou émergents sur le microbiome, soulignant les inexactitudes factuelles. Nous commençons par des points relativement mineurs, mais illustratifs, et progressons vers des problèmes ayant des impacts potentiels plus importants. Nous avons délibérément essayé d’éviter de pointer inutilement du doigt les sources originales d’informations erronées, et espérons plutôt que nos idées et notre évaluation critique seront utiles sur le terrain. points et s’orienter vers des problèmes ayant des impacts potentiels plus importants. Nous avons délibérément essayé d’éviter de pointer inutilement du doigt les sources originales d’informations erronées, et espérons plutôt que nos idées et notre évaluation critique seront utiles sur le terrain. points et s’orienter vers des problèmes ayant des impacts potentiels plus importants. Nous avons délibérément essayé d’éviter de pointer inutilement du doigt les sources originales d’informations erronées, et espérons plutôt que nos idées et notre évaluation critique seront utiles sur le terrain.
« La recherche sur le microbiome est un nouveau domaine »
Le rythme de la recherche sur le microbiome humain s’est considérablement accéléré au cours des 15 dernières années, mais le domaine n’en est pas à ses balbutiements. L’affirmer ne rend pas service à l’excellente recherche qui a précédé l’avènement des approches de séquençage d’ADN à haut débit. En effet, il y a eu une riche histoire de recherche sur les micro-organismes associés à l’homme depuis au moins la fin du XIXe siècle. Escherichia coli a été isolé pour la première fois en 1885 1 , les bifidobactéries ont été décrites en 1899 2 et Metchnikoff a spéculé sur l’importance des micro-organismes intestinaux bénéfiques au début des années 1900 3 . De même, des concepts tels que l’axe intestin-cerveau font l’objet de recherches depuis des siècles 4et les impacts sur la santé des principaux métabolites associés au microbiome, tels que les acides gras à chaîne courte, ont été signalés pour la première fois il y a plus de 40 ans 5 .
« Joshua Lederberg a inventé le terme ‘microbiome' »
Bien que le lauréat du prix Nobel Joshua Lederberg ait réalisé de nombreuses réalisations notables dans sa carrière, il n’a pas inventé le mot « microbiome ». Cette affirmation souvent répétée a été complètement réfutée ailleurs, avec des preuves fournies que le mot a été utilisé dans son contexte moderne plus d’une décennie avant que Joshua Lederberg ne l’utilise pour la première fois en 2001 6 . Bien que relativement trivial, ce mythe sert d’exemple de la facilité avec laquelle les mensonges s’incrustent dans la littérature sur le microbiome humain.
« Il y a 10 12 cellules bactériennes par gramme de fèces humaines »
Ce chiffre est couramment mentionné dans la littérature sur le microbiome, mais sa source a été difficile à déterminer. Cependant, il peut provenir du comptage des cellules fécales en poids sec plutôt qu’en poids humide. Quoi qu’il en soit, c’est incorrect. Le chiffre réel, déterminé à l’aide de diverses méthodes telles que le comptage direct des cellules, l’hybridation in situ par fluorescence, la cytométrie en flux et la réaction quantitative en chaîne par polymérase (qPCR), se situe généralement entre 10 10 et 10 11 cellules microbiennes par gramme de poids humide de fèces 7 , 8 , 9 .
« Le microbiote humain pèse 1 à 2 kg »
Bien que cela soit mentionné à plusieurs reprises dans la littérature, il est souvent donné sans citation et nous n’avons pas pu trouver de source originale pour cette affirmation. Néanmoins, il est peu probable que ce soit vrai dans la plupart des cas. La majorité du microbiote humain réside dans le côlon, et ces micro-organismes représentent généralement moins de la moitié du poids des solides fécaux 10 . Les selles humaines moyennes pèsent moins de 200 g (poids humide) 11 , avec un contenu colique total allant de 83 à 421 g dans une petite étude sur les victimes de mort subite 12. Par conséquent, mis à part peut-être de rares cas d’individus sévèrement constipés avec des matières fécales extrêmement compactées dans leurs côlons, le poids total du microbiote humain est beaucoup plus susceptible d’être inférieur à 500 g, et peut-être même considérablement inférieur à cela dans certains cas.
« Le microbiote dépasse les cellules humaines de 10:1 »
Ce mythe est peut-être l’un des plus répandus dans la littérature sur le microbiome humain et c’est celui que nous avons également répété sans critique dans le passé (nous ne sommes malheureusement pas à l’abri de la mythologie). D’excellents travaux 10 ont cependant montré que ce mythe semble provenir d’un calcul « au dos de l’enveloppe » dans les années 1970. Des analyses plus détaillées indiquent que le chiffre réel, bien que toujours impressionnant, est probablement plus proche d’un rapport de 1:1. Il convient de noter que le ratio est susceptible de varier d’une personne à l’autre et dépend de facteurs tels que la taille corporelle de l’hôte et la quantité de matières fécales qu’il transporte dans son côlon 13. Les estimations actuelles reposent également en grande partie sur les observations d’individus adultes vivant dans des pays urbanisés à revenu élevé. Des estimations plus complètes nécessiteront une étude des personnes issues de milieux ruraux ou à faible revenu, ainsi que de tout le parcours de vie.
« Le microbiote est hérité de la mère à la naissance »
Bien que des variantes de cette affirmation soient plus souvent trouvées dans les articles de vulgarisation scientifique que dans la littérature scientifique, c’est un exemple de l’extrême importance de la nuance dans la description du microbiome humain. Bien que certains micro-organismes soient directement transférés de la mère au bébé lors de la naissance 14 , 15 , proportionnellement peu d’espèces de microbiote sont véritablement « héréditaires » et persistent de la naissance à l’âge adulte chez la progéniture 15 , 16 . En effet, la majeure partie de l’expansion de la diversité du microbiote intestinal se produit après la naissance, au cours des premières années de vie, et augmente de façon plus spectaculaire après le sevrage 17 (Fig. 1). Chaque adulte se retrouve avec une configuration de microbiote unique, même des jumeaux identiques élevés dans le même foyer 18 . Par conséquent, bien que l’assemblage du microbiote ne soit pas encore entièrement compris, les communautés microbiennes adultes semblent être principalement façonnées par des expositions environnementales stochastiques antérieures, ainsi que par de multiples autres facteurs tels que l’alimentation, l’antibiothérapie et la génétique de l’hôte, avec un « héritage » direct de la mère à la naissance jouant un rôle similaire moindre.
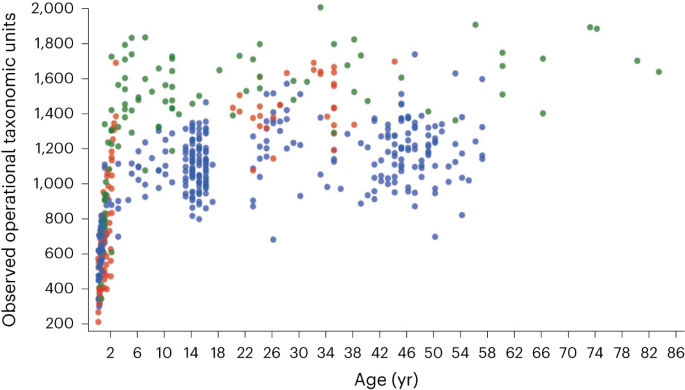 Fig. 1 : La diversité du microbiote intestinal humain augmente considérablement au cours des années suivant la naissance.
Fig. 1 : La diversité du microbiote intestinal humain augmente considérablement au cours des années suivant la naissance.« La plupart des maladies sont caractérisées par un pathobiome »
Il est devenu de plus en plus courant de lire dans la littérature des affirmations selon lesquelles la plupart des maladies sont causées par un « pathobiome ». Ceci est vaguement défini comme des interactions délétères entre les communautés microbiennes et leur hôte qui conduisent à la maladie. Ce terme est malheureusement trop simpliste et intrinsèquement erroné. Les micro-organismes et leurs métabolites ne sont ni « bons » ni « mauvais », ils existent simplement. Leurs impacts sur nous en tant qu’hôtes dépendent fortement du contexte. Les micro-organismes ou les métabolites qui sont nocifs dans un contexte peuvent ne causer aucun dommage dans un autre. Par exemple, Clostridioides difficile peut être porteur asymptomatique tout au long de la vie et ne causer des problèmes qu’à un âge avancé lorsque l’hôte est immunodéprimé et traité avec des antibiotiques 19 . De même, une souche d’ E. colipeut être relativement inoffensif dans le côlon, mais provoquer une infection des voies urinaires s’il envahit l’urètre 20 . En conséquence, le terme pathobiome reste vague et manque de la précision requise pour être vraiment utile en pratique clinique.
Il est vrai, cependant, qu’il a été démontré que de nombreuses conditions humaines sont en corrélation avec des altérations de la composition du microbiote. Ceci est parfois appelé « dysbiose », qui est également un terme vague avec une applicabilité clinique limitée 21 . Il est très probable que cela puisse contribuer à la progression de la maladie dans certaines conditions, y compris les maladies inflammatoires de l’intestin 22 , 23 , cependant, de telles altérations sont rarement cohérentes et le microbiote est extrêmement variable entre les individus, à la fois en santé et en maladie. Il est donc extrêmement difficile d’identifier les configurations du microbiote intestinal avec la spécificité et la reproductibilité requises pour la pratique clinique 24. De plus, corréler les changements du microbiome intestinal avec des marqueurs ou des données systémiques est semé d’embûches. Cela ne tient souvent pas compte des facteurs de confusion tels que l’âge, l’indice de masse corporelle (IMC), le sexe et les médicaments, des facteurs tels que les interactions avec la communauté microbienne ou les changements qui se produisent à la suite de changements immunologiques, métaboliques ou autres chez l’hôte plutôt que d’être directement causal (Fig. 2 ). Les tentatives visant à définir des «points de basculement» auxquels les changements dans la composition du microbiome influencent définitivement la progression de la maladie ont jusqu’à présent largement échoué à générer un consensus clair en raison d’un manque de cohérence entre les différentes études. Il s’agit donc d’un saut qui n’est pas encore fondé sur des preuves pour conclure qu’un pathobiome caractéristique joue un rôle dans « la plupart » des maladies.
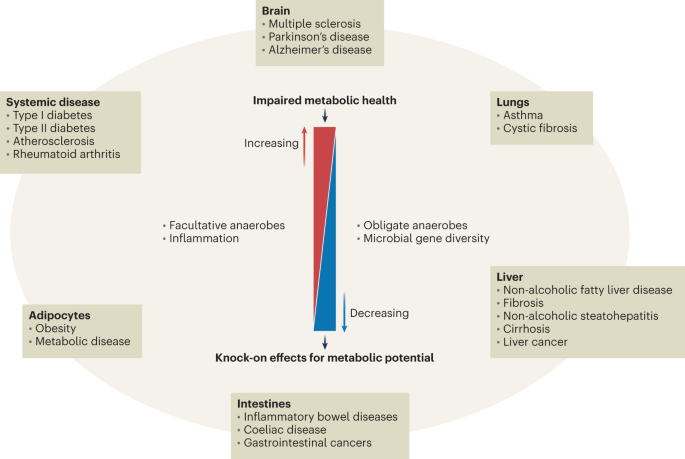 Fig. 2 : Difficultés d’établir la causalité à partir d’études de microbiote basées sur la corrélation.
Fig. 2 : Difficultés d’établir la causalité à partir d’études de microbiote basées sur la corrélation.« Le rapport Firmicutes:Bacteroidetes est altéré dans l’obésité »
Liée à la section précédente, cette affirmation couramment utilisée mais erronée découle principalement de la recherche basée sur les rongeurs et des résultats d’études humaines uniques ou sous-alimentées. Cependant, comme pour de nombreuses autres études qui rapportent des liens entre des profils de microbiote spécifiques et la maladie, la reproductibilité est médiocre. En effet, il y a maintenant eu au moins trois méta-analyses rapportant que cette découverte est incohérente entre les études humaines et qu’il n’y a, en fait, aucune signature taxonomique microbienne reproductible de l’obésité chez l’homme 25 , 26 , 27 .
Cette idée fausse reflète également une tendance inutile à examiner les profils de microbiote basés sur des séquences à des niveaux taxonomiques très larges, tels que les embranchements. Bien que cela soit attrayant du point de vue de la simplification des données, cela ne tient pas compte de l’énorme variabilité inhérente au sein des embranchements individuels. Pour faire une analogie grossière, les humains, les oiseaux, les poissons, les reptiles et même les ascidies sont tous membres du phylum Chordata, mais ont clairement des physiologies, des modes de vie et des impacts très différents sur leurs environnements.
De plus, cette affirmation était également basée sur des données de séquence d’ADN basées sur l’abondance relative. Les données de composition sont toujours utiles, et peuvent bien corréler avec les quantifications absolues obtenues avec des techniques telles que la qPCR 28 , 29 . Cependant, certaines études ont suggéré que les corrélations basées sur l’abondance relative peuvent perdre de leur importance lorsque les abondances microbiennes absolues sont également prises en compte dans les analyses 9 . À l’avenir, l’incorporation accrue de données de quantification absolue peut aider à rendre les conclusions basées sur des analyses de composition plus robustes.
« Le microbiome intestinal est fonctionnellement redondant »
Cette affirmation découle d’études montrant que, alors que la composition taxonomique des métagénomes humains peut varier énormément, les profils de prédiction des gènes fonctionnels restent remarquablement cohérents. Nous soutenons qu’il s’agit au moins en partie d’artefacts, car ces comparaisons fonctionnelles sont généralement effectuées après avoir rejeté la grande proportion de données métagénomiques qui ne correspondent pas aux bases de données de référence 30. Une grande partie de ce qui correspond à ces bases de données est probablement dérivée de gènes courants et/ou bien caractérisés, qui se trouvent dans de nombreuses bactéries différentes et sont également relativement bien représentés dans les bases de données de référence. Ces analyses comparatives ne permettent donc pas d’appréhender avec précision les fonctions spécialisées ou moins bien caractérisées. Ainsi, la vérité est plus nuancée. Bien qu’il existe des fonctionnalités importantes qui sont conservées dans de nombreuses espèces différentes de microbiote humain, telles que la production d’acides gras à chaîne courte 29 , il existe de nombreuses fonctions clés qui ne sont exercées que par un nombre relativement restreint d’espèces de microbiote. Les exemples incluent l’oxalate 31 et l’amidon résistant 32dégradation. En l’absence d’espèces clés, de telles fonctionnalités ne sont pas nécessairement entièrement remplacées par d’autres membres du microbiote.
« Le séquençage est impartial »
Bien que les méthodes basées sur les séquences aient transformé la recherche sur le microbiome, elles ne sont pas parfaites. Des biais peuvent être introduits à chaque étape des études basées sur la séquence, de la collecte et du stockage des échantillons, en passant par les étapes en laboratoire telles que l’extraction de l’ADN, jusqu’au choix des pipelines bioinformatiques et des bases de données de référence utilisées pour analyser les données 33 . Des comparaisons d’études du microbiote basées sur des séquences et sur des cultures ont montré que les approches basées sur des séquences échouaient complètement à détecter certaines espèces qui n’étaient récupérées qu’à l’aide de méthodes de culture traditionnelles 34 . Les approches modernes basées sur les séquences sont extrêmement puissantes mais, comme toutes les techniques, elles ne sont pas impartiales. Pour une interprétation optimale des résultats, il est important d’être conscient des limites inhérentes à toute méthode donnée.
« Nous avons besoin de méthodologies standardisées »
Cette opinion est répandue dans le domaine du microbiome et est raisonnablement fondée sur le désir de faciliter et de rendre plus robuste la comparaison des résultats de différentes études. Cependant, comme indiqué ci-dessus, aucune méthodologie n’est parfaite et toutes sont biaisées d’une manière ou d’une autre. Si tout le monde dans le monde utilise la même méthode, alors tout le monde est également aveugle aux limites de cette approche particulière. Il y a aussi le problème de décider quel protocole tout le monde devrait utiliser. Par exemple, les comparaisons des résultats du projet sur le microbiome humain avec le projet MetaHIT ont montré des différences marquées dans les profils de microbiome et ont indiqué que le protocole du projet sur le microbiome humain était moins efficace pour extraire l’ADN des eucaryotes et des lignées bactériennes à Gram positif 35. La vérité est que la « meilleure » méthode dépend fondamentalement de la structure sous-jacente de la communauté microbienne dans un échantillon donné et cela peut varier énormément entre les individus et entre les sites corporels. Pour ces raisons, nous soutenons, comme d’autres l’ont fait, que l’optimisation et la vérification des résultats basés sur la séquence avec des approches supplémentaires sont préférables à demander à tout le monde d’adopter la même méthode 36 . Un avantage supplémentaire des études à multiples facettes utilisant différentes méthodes et plates-formes de recherche est qu’elles peuvent permettre une compréhension plus mécaniste des associations entre les micro-organismes et les phénotypes hôtes 37 , 38 , 39. Une transparence accrue lors de la déclaration des choix de méthodologie serait utile pour comparer les résultats de différentes études. Les lignes directrices récemment publiées STORMS (Strengthening the Organization and Reporting of Microbiome Studies) 40 , par exemple, pourraient grandement faciliter ce processus si elles sont largement adoptées.
« La plupart du microbiote humain est ‘incultivable' »
L’adoption de technologies basées sur des séquences à haut débit a également été reflétée par des affirmations de certains milieux selon lesquelles ces méthodes doivent être utilisées car la plupart des micro-organismes associés à l’homme ne peuvent pas être cultivés en laboratoire. En effet, une proportion assez importante de la composante bactérienne et archéenne de notre microbiote est déjà cultivée 41 (virus et champignons restent sous-représentés), des travaux pionniers dès les années 1970 établissant la cultivabilité d’une grande diversité d’espèces le microbiote intestinal humain 42 . Beaucoup plus d’espèces continuent d’être cultivées à mesure que les efforts en laboratoire ont augmenté le débit 43 , 44. Cela implique que les lacunes actuelles dans les collections de cultures sont au moins en partie attribuables à un manque d’efforts antérieurs plutôt qu’à une « inculturabilité » inhérente. Bien que la culture soit indéniablement à forte intensité de main-d’œuvre, ait ses propres biais et nécessite souvent un équipement et des milieux spécialisés coûteux, il y a des avantages évidents à avoir des micro-organismes en culture. Il s’agit notamment de permettre des expériences mécanistes, de vérifier les prédictions génomiques et de les développer en tant que nouvelles thérapies 45. Compte tenu de l’importance de la poursuite des travaux basés sur la culture pour la progression de la recherche sur le microbiome, il est gratifiant que ce mythe soit devenu moins répandu ces dernières années suite à la publication des études à fort impact susmentionnées qui démontrent qu’il est faux. Cependant, cela constitue un excellent exemple de la façon dont un dogme précédemment largement accepté est parfois tout simplement faux. Il existe des leçons importantes pour de nombreux autres mythes et idées fausses qui n’ont pas encore été aussi largement rejetés.
conclusion
Le domaine du microbiome est vaste et de nombreux autres sujets controversés auraient également pu être inclus ici. Cependant, les connaissances évoluent encore sur bon nombre d’entre elles; par conséquent, nous nous sommes largement concentrés sur les concepts pour lesquels nous pensons qu’il existe une base solide de preuves pour rejeter les mythes et les idées fausses. Bien que certains des points ci-dessus puissent sembler triviaux, nous soutenons que l’exactitude de détails tels que ceux-ci est importante. Si nous répétons constamment des mensonges sur des détails mineurs, pouvons-nous compter sur notre exactitude pour couvrir des sujets plus importants ? Nous espérons qu’en illustrant seulement quelques exemples de mythes et d’idées fausses sur le microbiome, nous pourrons attirer davantage l’attention sur les problèmes potentiels de simplification excessive et d’évaluation critique insuffisante dans la littérature sur le microbiome.
Compte tenu des nombreux impacts potentiels sur la santé, de l’énorme montant de financement et du vif intérêt du public pour les microbiomes, le rejet des affirmations non fondées est crucial si nous souhaitons éviter de dépenser des ressources limitées pour rechercher des voies improductives et saper la confiance du public dans nos conclusions.